
Translation des DNA-Prozesses in Eukaryoten und Prokaryoten

Das DNA-Translation ist der Prozess, durch den die in den während der Transkription erzeugten Messenger-RNAs enthaltenen Informationen (die Kopie der Informationen in einer DNA-Sequenz in Form von RNA) mittels Proteinsynthese in eine Aminosäuresequenz "übersetzt" werden.
Aus zellulärer Sicht ist die Genexpression eine relativ komplexe Angelegenheit, die in zwei Schritten abläuft: Transkription und Translation..
Alle Gene, die exprimiert werden (unabhängig davon, ob sie Peptidsequenzen, dh Proteine, codieren oder nicht), tun dies zunächst, indem sie die in ihrer DNA-Sequenz enthaltenen Informationen durch einen als Transkription bezeichneten Prozess auf ein Messenger-RNA (mRNA) -Molekül übertragen.
Die Transkription wird durch spezielle Enzyme erreicht, die als RNA-Polymerasen bekannt sind und einen der komplementären Stränge der DNA des Gens als Matrize für die Synthese eines "Prä-mRNA" -Moleküls verwenden, das anschließend zu einer reifen mRNA verarbeitet wird..
Für Gene, die für Proteine kodieren, werden die in reifen mRNAs enthaltenen Informationen "gelesen" und gemäß dem genetischen Code, der angibt, welches Codon oder Nukleotidtriplett welcher bestimmten Aminosäure entspricht, in Aminosäuren übersetzt..
Die Spezifikation der Aminosäuresequenz eines Proteins hängt daher von der Anfangssequenz der stickstoffhaltigen Basen in der DNA ab, die dem Gen entspricht, und dann in der mRNA, die diese Information vom Kern zum Cytosol transportiert (in eukaryotischen Zellen); Prozess, der auch als mRNA-gesteuerte Proteinsynthese definiert wird.
Da es 64 mögliche Kombinationen der 4 stickstoffhaltigen Basen gibt, aus denen DNA und RNA bestehen, und nur 20 Aminosäuren, kann eine Aminosäure von verschiedenen Tripletts (Codons) codiert werden, weshalb der genetische Code als "entartet" bezeichnet wird. (mit Ausnahme der Aminosäure Methionin, die von einem einzigartigen AUG-Codon codiert wird).
Artikelverzeichnis
- 1 Eukaryotische Übersetzung (Schritt-Prozess)
- 1.1 - Verarbeitung von mRNAs vor ihrer Translation
- 1.2 - Ribosomen
- 1.3 - Übersetzung beginnen
- 2 Prokaryontische Übersetzung (Schritte-Prozesse)
- 2.1 Prozess
- 3 Referenzen
Eukaryotische Übersetzung (Schritt-Prozess)
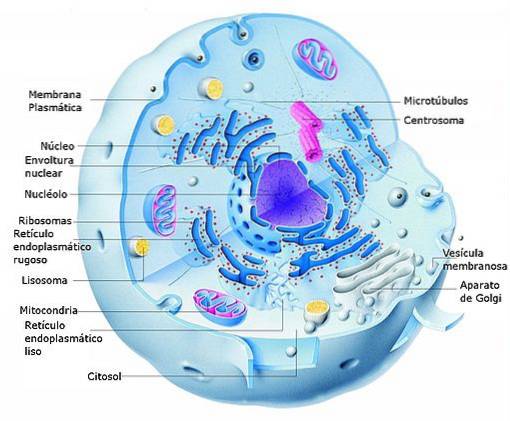
In eukaryotischen Zellen findet die Transkription im Zellkern und die Translation im Zytosol statt, sodass die beim ersten Prozess gebildeten mRNAs auch beim Informationstransport vom Zellkern zum Zytosol eine Rolle spielen, wo sich die Zellen befinden. Biosynthesemaschinerie (Ribosomen).
Es ist wichtig zu erwähnen, dass die Kompartimentierung von Transkription und Translation in Eukaryoten für den Kern gilt, nicht jedoch für Organellen mit eigenem Genom wie Chloroplasten und Mitochondrien, deren Systeme denen prokaryotischer Organismen ähnlicher sind..
Eukaryontische Zellen haben auch cytosolische Ribosomen, die an die Membranen des endoplasmatischen Retikulums gebunden sind (raues endoplasmatisches Retikulum), bei denen die Translation von Proteinen erfolgt, die zur Insertion in Zellmembranen bestimmt sind oder die eine posttranslationale Verarbeitung erfordern, die in diesem Kompartiment stattfindet..
- Verarbeitung von mRNAs vor ihrer Translation
Die mRNAs werden an ihren Enden modifiziert, wenn sie transkribiert werden:
- Wenn das 5'-Ende der mRNA während der Transkription aus der Oberfläche der RNA-Polymerase II austritt, wird sie sofort von einer Gruppe von Enzymen "angegriffen", die eine "Haube" synthetisieren, die aus 7-Methylguanylat besteht und mit dem terminalen Nukleotid verbunden ist der mRNA über eine 5 ', 5' Triphosphatbindung.
- Das 3'-Ende der mRNA unterliegt einer "Spaltung" durch eine Endonuklease, die eine 3'-freie Hydroxylgruppe erzeugt, an die eine "Kette" oder ein "Schwanz" von Adeninresten (von 100 bis 250) gebunden ist, denen eine hinzugefügt wird gleichzeitig durch ein Enzym poly (A) Polymerase.
Die "Haube 5 '" und der "Schwanz poly A ”erfüllen Funktionen beim Schutz von mRNA-Molekülen vor Abbau und zusätzlich beim Transport reifer Transkripte zum Cytosol bzw. bei der Initiierung und Beendigung der Translation..
C.orte und spleißen
Nach der Transkription durchlaufen die "primären" mRNAs mit ihren beiden noch im Kern vorhandenen modifizierten Enden einen "Spleiß" -Prozess, bei dem im Allgemeinen intronische Sequenzen entfernt und die resultierenden Exons verbunden werden (posttranskriptionelle Verarbeitung), mit denen sich reife Transkripte befinden erhalten, die den Kern verlassen und das Cytosol erreichen.
Das Spleißen erfolgt durch einen Riboproteinkomplex namens Spleißosom (Anglizismus von Spleißosom), bestehend aus fünf kleinen Ribonukleoproteinen und RNA-Molekülen, die in der Lage sind, die aus dem Primärtranskript zu entfernenden Regionen zu „erkennen“.
In vielen Eukaryoten gibt es ein Phänomen, das als "alternatives Spleißen" bekannt ist, was bedeutet, dass verschiedene Arten von posttranskriptionellen Modifikationen verschiedene Proteine oder Isoenzyme erzeugen können, die sich in einigen Aspekten ihrer Sequenzen voneinander unterscheiden..
- Ribosomen
Wenn reife Transkripte den Kern verlassen und zur Translation in das Cytosol transportiert werden, werden sie von dem als Ribosom bekannten Translationskomplex verarbeitet, der aus einem Komplex von Proteinen besteht, die mit RNA-Molekülen assoziiert sind..
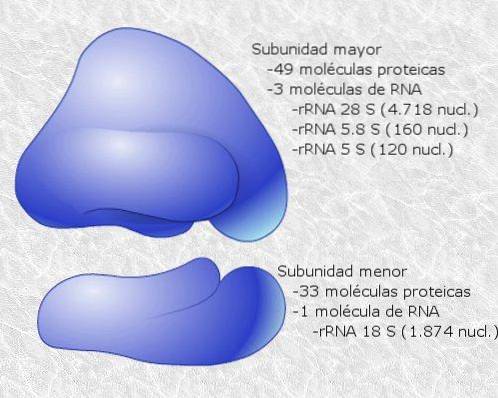
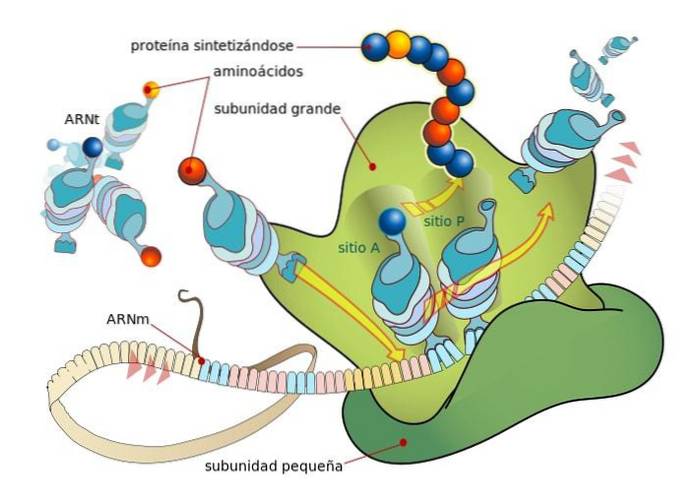
Ribosomen bestehen aus zwei Untereinheiten, einer "großen" und einer "kleinen", die im Cytosol frei dissoziiert sind und an das translatierte mRNA-Molekül binden oder assoziieren.
Die Bindung zwischen Ribosomen und mRNA hängt von spezialisierten RNA-Molekülen ab, die mit ribosomalen Proteinen (ribosomale RNA oder rRNA und Transfer-RNA oder tRNA) assoziieren, von denen jedes spezifische Funktionen hat..
TRNAs sind molekulare "Adapter", da sie durch eines ihrer Enden jedes Codon oder Triplett in der reifen mRNA "lesen" können (durch Basenkomplementarität) und durch das andere an die Aminosäure binden können, die vom "gelesenen" Codon codiert wird..
Andererseits sind die rRNA-Moleküle für die Beschleunigung (Katalyse) des Bindungsprozesses jeder Aminosäure in der entstehenden Peptidkette verantwortlich.
Eine reife eukaryotische mRNA kann von vielen Ribosomen "gelesen" werden, so oft die Zelle dies anzeigt. Mit anderen Worten, dieselbe mRNA kann zu vielen Kopien desselben Proteins führen..
Codon starten und Frame lesen
Wenn eine reife mRNA von ribosomalen Untereinheiten angefahren wird, "scannt" der Riboproteinkomplex die Sequenz des Moleküls, bis er ein Startcodon findet, das immer AUG ist und die Einführung eines Methioninrests beinhaltet.
Das AUG-Codon definiert den Leserahmen für jedes Gen und definiert zusätzlich die erste Aminosäure aller in der Natur translatierten Proteine (diese Aminosäure wird häufig posttranslational eliminiert)..
Codons stoppen
Drei weitere Codons wurden als solche identifiziert, die eine Translationsterminierung induzieren: UAA, UAG und UGA..
Diejenigen Mutationen, die eine Änderung der stickstoffhaltigen Basen im Triplett beinhalten, das für eine Aminosäure kodiert, und die zu Stopcodons führen, werden als Nonsense-Mutationen bezeichnet, da sie einen vorzeitigen Stillstand des Synthesevorgangs verursachen, der kürzere Proteine bildet.
Nicht übersetzte Regionen
Nahe dem 5'-Ende reifer mRNA-Moleküle gibt es Regionen, die nicht translatiert sind (UTR). Nicht übersetzte Region), auch "Leader" -Sequenzen genannt, die sich zwischen dem ersten Nukleotid und dem Translationsstartcodon (AUG) befinden.
Diese UTR-Regionen, die nicht translatiert sind, haben spezifische Stellen für die Bindung an Ribosomen und beispielsweise beim Menschen eine Länge von ungefähr 170 Nukleotiden, darunter regulatorische Regionen, Proteinbindungsstellen, die bei der Regulation der Translation usw..
- Beginn der Übersetzung
Die Translation sowie die Transkription bestehen aus drei Phasen: einer Initiationsphase, einer Elongationsphase und schließlich einer Terminationsphase..
Einleitung
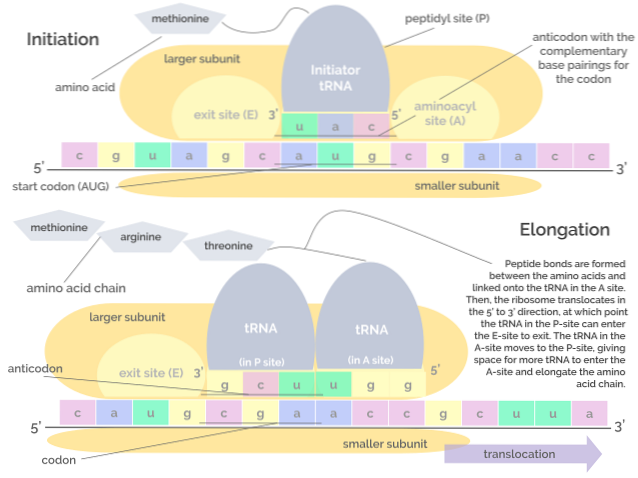
Es besteht aus dem Aufbau des Translationskomplexes auf der mRNA, der die Vereinigung von drei Proteinen verdient, die als Initiationsfaktoren (IF) bekannt sind. Initiationsfaktor) IF1, IF2 und IF3 zur kleinen Untereinheit des Ribosoms.
Der durch die Initiationsfaktoren und die kleine ribosomale Untereinheit gebildete "Vorinitiations" -Komplex bindet wiederum an eine tRNA, die einen Methioninrest "trägt", und dieser Satz von Molekülen bindet an die mRNA nahe dem Startcodon. AUG.
Diese Ereignisse führen zur Bindung der mRNA an die große ribosomale Untereinheit, was zur Freisetzung von Initiationsfaktoren führt. Die große Ribosomenuntereinheit hat 3 Bindungsstellen für tRNA-Moleküle: die A-Stelle (Aminosäure), die P-Stelle (Polypeptid) und die E-Stelle (Ausgang)..
Stelle A bindet an das Anticodon der Aminoacyl-tRNA, das zu dem der zu translatierenden mRNA komplementär ist; An der P-Stelle wird die Aminosäure von der tRNA auf das entstehende Peptid übertragen, und an der E-Stelle befindet sie sich in der "leeren" tRNA, bevor sie nach Abgabe der Aminosäure in das Cytosol freigesetzt wird.
Verlängerung
Diese Phase besteht aus der "Bewegung" des Ribosoms entlang des mRNA-Moleküls und der Translation jedes Codons, das "liest", was das Wachstum oder die Verlängerung der Polypeptidkette bei der Geburt impliziert..
Dieser Prozess erfordert einen Faktor, der als Elongationsfaktor G bekannt ist, und Energie in Form von GTP, was die Translokation von Elongationsfaktoren entlang des mRNA-Moleküls während der Translation antreibt..
Die Peptidyltransferaseaktivität von ribosomalen RNAs ermöglicht die Bildung von Peptidbindungen zwischen aufeinanderfolgenden Aminosäuren, die der Kette hinzugefügt werden.
Beendigung
Die Translation endet, wenn das Ribosom auf eines der Terminationscodons trifft, da tRNAs diese Codons nicht erkennen (sie codieren keine Aminosäuren). Proteine, die als Freisetzungsfaktoren bekannt sind, binden ebenfalls, was die Ablösung von mRNA vom Ribosom und die Dissoziation seiner Untereinheiten erleichtert..
Prokaryotische Übersetzung (Schritte-Prozesse)
In Prokaryoten wie in eukaryotischen Zellen befinden sich die für die Proteinsynthese verantwortlichen Ribosomen im Cytosol (was auch für die Transkriptionsmaschinerie gilt), was eine schnelle Erhöhung der cytosolischen Konzentration eines Proteins bei der Expression der Gene ermöglicht diese Kodierung nimmt zu.
Obwohl dies bei diesen Organismen kein äußerst häufiger Prozess ist, können die während der Transkription produzierten primären mRNAs durch "Spleißen" eine posttranskriptionelle Reifung erfahren. Am häufigsten werden jedoch Ribosomen beobachtet, die an das Primärtranskript gebunden sind und es gleichzeitig mit der Transkription aus der entsprechenden DNA-Sequenz translatieren..
In Anbetracht des Vorstehenden beginnt die Translation in vielen Prokaryoten am 5'-Ende, da das 3'-Ende der mRNA an der Matrizen-DNA gebunden bleibt (und gleichzeitig mit der Transkription auftritt)..
Nicht übersetzte Regionen
Prokaryontische Zellen produzieren auch mRNA mit nicht translatierten Regionen, die als "Shine-Dalgarno-Box" bekannt sind und deren Konsensussequenz AGGAGG ist. Es ist offensichtlich, dass die UTR-Regionen von Bakterien erheblich kürzer sind als die von eukaryotischen Zellen, obwohl sie während der Translation ähnliche Funktionen ausüben..
Prozess
In Bakterien und anderen prokaryotischen Organismen ist der Translationsprozess dem von eukaryotischen Zellen ziemlich ähnlich. Es besteht auch aus drei Phasen: Initiierung, Verlängerung und Beendigung, die von spezifischen prokaryotischen Faktoren abhängen, die sich von den von Eukaryoten verwendeten unterscheiden..
Die Dehnung hängt beispielsweise eher von den als EF-Tu und EF-Ts bekannten Dehnungsfaktoren als vom G-Faktor der Eukaryoten ab..
Verweise
- B. Alberts, A. Johnson, J. Lewis, M. Raff, K. Roberts & P. Walter (2007). Molekularbiologie der Zelle. Garland Science. New York, 1392.
- Clancy, S. & Brown, W. (2008) Übersetzung: DNA zu mRNA zu Protein. Naturerziehung 1 (1): 101.
- A. J. Griffiths, S. R. Wessler, R. C. Lewontin, W. M. Gelbart, D. T. Suzuki & J. H. Miller (2005). Eine Einführung in die genetische Analyse. Macmillan.
- H. Lodish, A. Berk, C. A. Kaiser, M. Krieger, M. P. Scott, A. Bretscher,… & P. Matsudaira (2008). Molekulare Zellbiologie. Macmillan.
- Nelson, D. L., Lehninger, A. L. & Cox, M. M. (2008). Lehninger Prinzipien der Biochemie. Macmillan.
- Rosenberg, L. E. & Rosenberg, D. D. (2012). Menschliche Gene und Genome: Wissenschaft. Gesundheit, Gesellschaft, 317-338.



Bisher hat noch niemand einen Kommentar zu diesem Artikel abgegeben.