
Struktur, Klassifizierung, Funktion und Bedeutung der Zinkfinger
Das Zinkfinger (ZF) sind Strukturmotive, die in einer großen Anzahl von eukaryotischen Proteinen vorhanden sind. Sie gehören zur Gruppe der Metalloproteine, da sie das Zinkmetallion binden können, das sie für ihren Betrieb benötigen. Es wird vorausgesagt, dass mehr als 1.500 ZF-Domänen in rund 1.000 verschiedenen Proteinen beim Menschen existieren..
Der Begriff Zinkfinger oder "Zinkfinger" wurde erstmals 1985 von Miller, McLachlan und Klug geprägt, während die kleinen DNA-Bindungsdomänen des Transkriptionsfaktors TFIIIA von im Detail untersucht wurden Xenopus laevis, einige Jahre zuvor von anderen Autoren beschrieben.
Proteine mit ZF-Motiven gehören zu den am häufigsten vorkommenden Proteinen im Genom eukaryotischer Organismen und sind an einer Vielzahl essentieller zellulärer Prozesse beteiligt, darunter Gentranskription, Proteintranslation, Metabolismus, Faltung und Assemblierung anderer Proteine und Lipide Andere.
Artikelverzeichnis
- 1 Struktur
- 2 Klassifizierung
- 2.1 C2H2
- 2.2 C2H
- 2.3 C4 (Bogen oder Band)
- 2.4 C4 (GATA-Familie)
- 2,5 C6
- 2.6 Zinkfinger (C3HC4-C3H2C3)
- 2,7 H2C2
- 3 Funktionen
- 4 Biotechnologische Bedeutung
- 5 Referenzen
Struktur
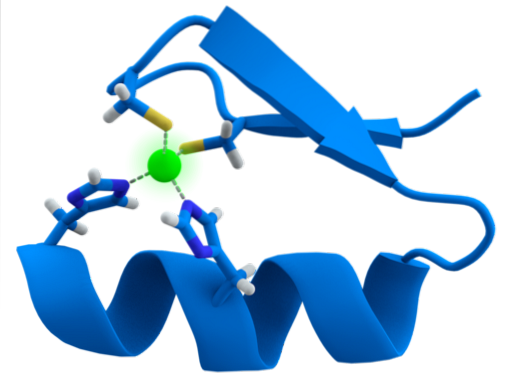
Die Struktur der ZF-Motive ist äußerst konserviert. Normalerweise haben diese wiederholten Regionen 30 bis 60 Aminosäuren, deren Sekundärstruktur als zwei antiparallele Beta-Blätter vorliegt, die eine Haarnadel und eine Alpha-Helix bilden, die als ββα bezeichnet wird.
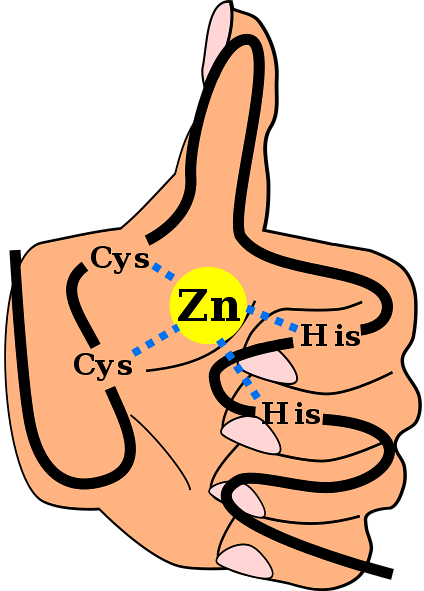
Diese Sekundärstruktur wird durch hydrophobe Wechselwirkungen und durch die Koordination eines Zinkatoms stabilisiert, das durch zwei Cystein- und zwei Histidinreste (Cys) gegeben istzweiSeinezwei). Es gibt jedoch ZFs, die mehr als ein Zinkatom koordinieren können, und andere, bei denen die Reihenfolge der Cys- und His-Reste variiert..
Die ZFs können in Chargen wiederholt gefunden werden, die linear im gleichen Protein konfiguriert sind. Alle haben ähnliche Strukturen, können jedoch durch Variationen von Aminosäureresten, die für die Erfüllung ihrer Funktionen entscheidend sind, chemisch voneinander unterschieden werden.
Ein gemeinsames Merkmal von ZFs ist ihre Fähigkeit, DNA- oder RNA-Moleküle unterschiedlicher Länge zu erkennen, weshalb sie ursprünglich nur als Transkriptionsfaktoren betrachtet wurden.
Im Allgemeinen werden 3bp-Regionen in der DNA erkannt und erreicht, wenn das Protein mit der ZF-Domäne die Alpha-Helix in der Hauptrille des DNA-Moleküls präsentiert.
Einstufung
Es gibt verschiedene ZF-Motive, die sich durch ihre Natur und die unterschiedlichen räumlichen Konfigurationen, die durch die Koordinationsbindungen mit dem Zinkatom erreicht werden, voneinander unterscheiden. Eine der Klassifikationen lautet wie folgt:
C.zweiH.zwei
Dies ist ein häufig vorkommendes Motiv in ZFs. Die meisten C-MotivezweiH.zwei Sie sind spezifisch für die Interaktion mit DNA und RNA, es wurde jedoch beobachtet, dass sie an Protein-Protein-Interaktionen beteiligt sind. Sie haben zwischen 25 und 30 Aminosäurereste und gehören zur größten Familie der regulatorischen Proteine in Säugetierzellen.
C.zweiH.
Sie interagieren mit RNA und einigen anderen Proteinen. Sie werden hauptsächlich als Teil einiger Retrovirus-Kapsidproteine beobachtet und unterstützen die Verpackung von viraler RNA unmittelbar nach der Replikation..
C.4 (Bogen oder Band)
Proteine mit diesem Motiv sind Enzyme, die für die DNA-Replikation und -Transkription verantwortlich sind. Ein gutes Beispiel hierfür können die groben Enzyme der Phagen T4 und T7 sein.
C.4 (GATA-Familie)
Diese ZF-Familie umfasst Transkriptionsfaktoren, die die Expression wichtiger Gene in zahlreichen Geweben während der Zellentwicklung regulieren. Die Faktoren GATA-2 und 3 sind beispielsweise an der Hämatopoese beteiligt.
C.6
Diese Domänen sind typisch für Hefen, insbesondere das GAL4-Protein, das die Transkription von Genen aktiviert, die an der Verwendung von Galactose und Melibiose beteiligt sind..
Zinkfinger (C.3HC4-C.3H.zweiC.3)
Diese speziellen Strukturen besitzen 2 Subtypen von ZF-Domänen (C.3HC4 und C3H.zweiC.3) und sind in zahlreichen tierischen und pflanzlichen Proteinen enthalten.
Sie kommen in Proteinen wie RAD5 vor, die an der DNA-Reparatur in eukaryotischen Organismen beteiligt sind. Sie sind auch in RAG1 enthalten, das für die Rekonfiguration von Immunglobulinen essentiell ist.
H.zweiC.zwei
Diese Domäne von ZF ist in den Integrasen von Retroviren und Retrotransposons hoch konserviert; Durch die Bindung an das Zielprotein bewirkt es eine Konformationsänderung.
Eigenschaften
Proteine mit ZF-Domänen dienen verschiedenen Zwecken: Sie können in ribosomalen Proteinen oder in Transkriptionsadaptern gefunden werden. Sie wurden auch als integraler Bestandteil der Struktur der Hefe-RNA-Polymerase II nachgewiesen..
Sie scheinen an der intrazellulären Zinkhomöostase und an der Regulation der Apoptose oder des programmierten Zelltods beteiligt zu sein. Darüber hinaus gibt es einige Proteine mit ZF, die als Chaperone für die Faltung oder den Transport anderer Proteine fungieren..
Die Bindung an Lipide und eine grundlegende Rolle bei Protein-Protein-Wechselwirkungen sind ebenfalls wichtige Funktionen der ZF-Domänen in einigen Proteinen..
Biotechnologische Bedeutung
Im Laufe der Jahre hat das strukturelle und funktionelle Verständnis der ZF-Domänen große wissenschaftliche Fortschritte ermöglicht, bei denen ihre Eigenschaften für biotechnologische Zwecke genutzt werden..
Da einige Proteine mit ZF eine hohe Spezifität für bestimmte DNA-Domänen aufweisen, wird derzeit viel Aufwand in das Design spezifischer ZFs investiert, die wertvolle Fortschritte in der Gentherapie beim Menschen bringen können.
Interessante biotechnologische Anwendungen ergeben sich auch aus dem Design von Proteinen mit gentechnisch veränderten ZFs. Abhängig vom gewünschten Zweck können einige davon durch Zugabe von "Polyzink" -Fingerpeptiden modifiziert werden, die praktisch jede DNA-Sequenz mit hoher Affinität und Spezifität erkennen können..
Nuklease-modifizierte genomische Bearbeitung ist heute eine der vielversprechendsten Anwendungen. Diese Art der Bearbeitung bietet die Möglichkeit, Studien zur genetischen Funktion direkt im interessierenden Modellsystem durchzuführen..
Die Gentechnik mit modifizierten ZF-Nukleasen hat die Aufmerksamkeit von Wissenschaftlern auf dem Gebiet der genetischen Verbesserung von Pflanzensorten von agronomischer Bedeutung auf sich gezogen. Diese Nukleasen wurden verwendet, um ein endogenes Gen zu korrigieren, das in Tabakpflanzen herbizidresistente Formen erzeugt..
Nukleasen mit ZF wurden auch zur Zugabe von Genen in Säugetierzellen verwendet. Die fraglichen Proteine wurden verwendet, um einen Satz isogener Mauszellen mit einer Reihe von Allelen zu erzeugen, die für ein endogenes Gen definiert sind..
Ein solches Verfahren findet eine direkte Anwendung bei der Markierung und Schaffung neuer Allelformen, um die Beziehungen von Struktur und Funktion unter nativen Expressionsbedingungen und in isogenen Umgebungen zu untersuchen..
Verweise
- Berg, J. M. (1990). Zinkfinger-Domänen: Hypothesen und aktuelles Wissen. Jahresrückblick auf Biophysik und biophysikalische Chemie, 19(39), 405 & ndash; 421.
- B. Dreier, R. Beerli, D. Segal, J. Flippin & C. Barbas (2001). Entwicklung von Zinkfinger-Domänen zur Erkennung der 5'-ANN-3'-Familie von DNA-Sequenzen und deren Verwendung bei der Konstruktion künstlicher Transkriptionsfaktoren. JBC, (54).
- R. Gamsjäger, C. K. Liew, F. E. Loughlin, M. Crossley & J. P. Mackay (2007). Klebrige Finger: Zinkfinger als Proteinerkennungsmotive. Trends in den biochemischen Wissenschaften, 32(2), 63-70.
- Klug, A. (2010). Die Entdeckung von Zinkfingern und ihre Anwendungen in der Genregulation und Genommanipulation. Jahresrückblick Biochemie, 79(1), 213 & ndash; 231.
- K. Kluska, J. Adamczyk & A. Krȩzel (2017). Metallbindungseigenschaften von Zinkfingern mit einer natürlich veränderten Metallbindungsstelle. Metallomics, 10(2), 248 & ndash; 263.
- J. H. Laity, B. M. Lee & P. E. Wright (2001). Zinkfingerproteine: Neue Einblicke in die strukturelle und funktionelle Vielfalt. Aktuelle Meinung in der Strukturbiologie, elf(1), 39-46.
- J. Miller, A. D. McLachlan & A. Klug (1985). Repetitive Zinkbindungsdomänen im Proteintranskriptionsfaktor IIIA aus Xenopus-Oozyten. Zeitschrift für Spurenelemente in der experimentellen Medizin, 4(6), 1609 & ndash; 1614.
- F. D. Urnov, E. J. Rebar, M. C. Holmes, H. S. Zhang & P. D. Gregory (2010). Genom-Editing mit manipulierten Zinkfinger-Nukleasen. Nature Reviews Genetics, elf(9), 636-646.



Bisher hat noch niemand einen Kommentar zu diesem Artikel abgegeben.