
Anticodon
Was ist ein Anticodon??
EIN Anticodon ist eine Sequenz von drei Nukleotiden, die in einem Molekül der Transfer-RNA (tRNA) vorhanden ist, deren Funktion darin besteht, eine andere Sequenz von drei Nukleotiden zu erkennen, die in einem Molekül der Messenger-RNA (mRNA) vorhanden ist..
Diese Erkennung zwischen Codons und Anticodons ist antiparallel; Das heißt, einer befindet sich in der Richtung 5 '-> 3', während der andere in der Richtung 3 '-> 5' gekoppelt ist. Diese Erkennung zwischen drei Nukleotidsequenzen (Tripletts) ist für den Translationsprozess wesentlich; das heißt, bei der Synthese von Proteinen im Ribosom.
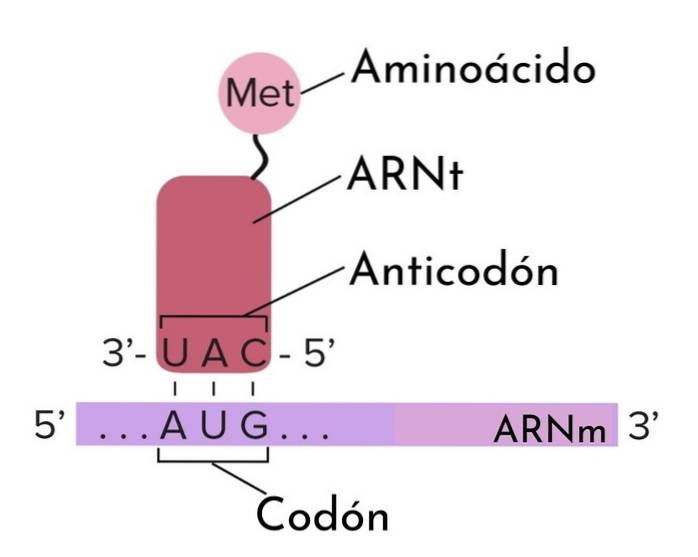
Während der Translation werden die Messenger-RNA-Moleküle durch die Erkennung ihrer Codons durch die Anticodons der Transfer-RNAs "gelesen". Diese Moleküle werden so genannt, weil sie eine bestimmte Aminosäure auf das Proteinmolekül übertragen, das auf dem Ribosom gebildet wird..
Es gibt 20 Aminosäuren, die jeweils von einem bestimmten Triplett codiert werden. Einige Aminosäuren werden jedoch von mehr als einem Triplett codiert..
Zusätzlich werden einige Codons von Anticodons in Transfer-RNA-Molekülen erkannt, an die keine Aminosäuren gebunden sind. Dies sind die sogenannten Stopcodons.
Beschreibung
Ein Anticodon besteht aus einer Sequenz von drei Nukleotiden, die eine der folgenden stickstoffhaltigen Basen enthalten können: Adenin (A), Guanin (G), Uracil (U) oder Cytosin (C) in einer Kombination von drei Nukleotiden, so dass dies funktioniert wie ein Code.
Anticodons kommen immer in Transfer-RNA-Molekülen vor und befinden sich immer 3 '-> 5'. Die Struktur dieser tRNAs ähnelt einem Klee, so dass er in vier Schleifen (oder Schleifen) unterteilt ist. In einer der Schleifen befindet sich das Anticodon.
Anticodons sind essentiell für die Erkennung von Messenger-RNA-Codons und folglich für den Proteinsynthesevorgang in allen lebenden Zellen.
Anticodon-Funktionen
Die Hauptfunktion von Anticodons ist die spezifische Erkennung von Tripletts, die Codons in Messenger-RNA-Molekülen bilden. Diese Codons sind die Anweisungen, die von einem DNA-Molekül kopiert wurden, um die Reihenfolge der Aminosäuren in einem Protein zu bestimmen..
Da die Transkription (die Synthese von Kopien der Messenger-RNA) in der 5'-> 3'-Richtung erfolgt, haben die Codons der Messenger-RNA diese Orientierung. Daher müssen die in den Transfer-RNA-Molekülen vorhandenen Anticodons die entgegengesetzte Orientierung haben, 3 '-> 5'..
Diese Vereinigung beruht auf Komplementarität. Wenn beispielsweise ein Codon 5'-AGG-3 'ist, ist das Anticodon 3'-UCC-5'. Diese Art der spezifischen Wechselwirkung zwischen Codons und Anticodons ist ein wichtiger Schritt, der es der Nukleotidsequenz in Messenger-RNA ermöglicht, eine Aminosäuresequenz innerhalb eines Proteins zu codieren..
Unterschiede zwischen Anticodon und Codon
- Anticodons sind Trinukleotideinheiten in tRNAs, die zu Codons in mRNAs komplementär sind. Sie ermöglichen es tRNAs, während der Proteinproduktion die richtigen Aminosäuren zu liefern. Stattdessen sind Codons Trinukleotideinheiten in DNA oder mRNA, die eine bestimmte Aminosäure in der Proteinsynthese codieren..
- Anticodons sind die Verbindung zwischen der Nukleotidsequenz der mRNA und der Aminosäuresequenz des Proteins. Codons übertragen vielmehr genetische Informationen vom Kern, in dem sich DNA befindet, auf Ribosomen, in denen die Proteinsynthese stattfindet..
- Das Anticodon befindet sich im Anticodon-Arm des tRNA-Moleküls im Gegensatz zu Codons, die sich im DNA- und mRNA-Molekül befinden..
- Das Anticodon ist komplementär zum jeweiligen Codon. Stattdessen ist das Codon in der mRNA komplementär zu einem Nukleotidtriplett eines bestimmten Gens in der DNA..
- Eine tRNA enthält ein Anticodon. Im Gegensatz dazu enthält eine mRNA eine Anzahl von Codons.
Die Swing-Hypothese
Die Swing-Hypothese schlägt vor, dass die Übergänge zwischen dem dritten Nukleotid des Codons der Messenger-RNA und dem ersten Nukleotid des Anticodons der Transfer-RNA weniger spezifisch sind als die Übergänge zwischen den beiden anderen Nukleotiden des Tripletts..
Crick beschrieb dieses Phänomen als "Schaukeln" an der dritten Position jedes Codons. In dieser Position passiert etwas, das es den Gelenken ermöglicht, weniger streng als normal zu sein. Es ist auch als Wackeln oder Wackeln bekannt.
Diese Crick-Wobble-Hypothese erklärt, wie sich das Anticodon einer bestimmten tRNA mit zwei oder drei verschiedenen mRNA-Codons paaren kann..
Crick schlug vor, dass, da die Basenpaarung (zwischen der Base 59 des Anticodons in tRNA und der Base 39 des Codons in mRNA) weniger streng als normal ist, an dieser Stelle ein gewisses "Wackeln" oder eine verringerte Affinität zulässig ist..
Infolgedessen erkennt eine einzelne tRNA häufig zwei oder drei der verwandten Codons, die eine bestimmte Aminosäure spezifizieren..
Normalerweise folgen Wasserstoffbrücken zwischen den Basen von tRNA-Anticodons und mRNA-Codons strengen Basenpaarungsregeln nur für die ersten beiden Basen des Codons. Dieser Effekt tritt jedoch nicht in allen dritten Positionen aller mRNA-Codons auf..
RNA und Aminosäuren
Basierend auf der Wobble-Hypothese wurde die Existenz von mindestens zwei Transfer-RNAs für jede Aminosäure mit Codons vorhergesagt, die eine vollständige Entartung aufweisen, was sich als wahr erwiesen hat..
Diese Hypothese sagte auch das Auftreten von drei Transfer-RNAs für die sechs Serin-Codons voraus. In der Tat wurden drei tRNAs für Serin charakterisiert:
- TRNA für Serin 1 (Anticodon AGG) bindet an die Codons UCU und UCC.
- TRNA für Serin 2 (AGU-Anticodon) bindet an die Codons UCA und UCG.
- TRNA für Serin 3 (Anticodon UCG) bindet an die Codons AGU und AGC.
Diese Spezifitäten wurden durch die stimulierte Bindung von gereinigten Aminoacyl-tRNA-Trinukleotiden an Ribosomen in vitro verifiziert..
Schließlich enthalten mehrere Transfer-RNAs die Base Inosin, die aus Purinhypoxanthin hergestellt wird. Inosin wird durch eine posttranskriptionelle Modifikation von Adenosin hergestellt.
Cricks Wobble-Hypothese sagte voraus, dass Inosin, wenn es am 5'-Ende eines Anticodons (der Wobble-Position) vorhanden ist, sich am Codon mit Uracil, Cytosin oder Adenin paaren würde.
Tatsächlich bindet gereinigte Alanyl-tRNA, die Inosin (I) an der 5'-Position des Anticodons enthält, an GCU-, GCC- oder GCA-Trinukleotid-aktivierte Ribosomen..
Das gleiche Ergebnis wurde mit anderen mit Inosin gereinigten tRNAs in der 5'-Position des Anticodons erhalten. Daher erklärt die Crick-Wobble-Hypothese sehr gut die Beziehungen zwischen tRNAs und Codons angesichts des genetischen Codes, der entartet, aber geordnet ist..
Verweise
- Brooker, R. (2012). Konzepte der Genetik (1. Aufl.). Die McGraw-Hill Companies, Inc..
- Brown, T. (2006). Genome 3 (3rd). Girlandenwissenschaft.
- Griffiths, A., Wessler, S., Carroll, S. & Doebley, J. (2015). Einführung in die genetische Analyse (11. Aufl.). W.H. Freeman
- Lewis, R. (2015). Humangenetik: Konzepte und Anwendungen(11. Aufl.). McGraw-Hill-Ausbildung.
- Snustad, D. & Simmons, M. (2011). Prinzipien der Genetik(6. Aufl.). John Wiley und Söhne.



Bisher hat noch niemand einen Kommentar zu diesem Artikel abgegeben.